En la película Jurassic Park se nos muestran los resultados de un experimento de ciencia ficción que intenta resucitar especies de dinosaurios extinguidos hace millones de años, a partir del DNA que habría quedado capturado e indemne dentro del interior de mosquitos atrapados en ámbar. Los dinosaurios forman parte de nuestra iconografía, probablemente porque desde niños nos sentimos atraídos inexplicablemente por su apariencia. Una de las grandes atracciones de muchos museos de historia natural son los esqueletos fósiles de dinosaurio que exponen. ¿Qué hay más imponente que el esqueleto fósil de tiranosaurio en el museo de Nueva York, que ha protagonizado varias películas, o el apatosaurus del Museo Británico de Historia Natural? Sin embargo, siempre que pensamos en fósiles, lo hacemos a gran escala, macroscópica, y raramente pensamos en que la vida en sus inicios era muy variada, y que muchas de las respuestas del futuro las tenemos que buscar en organismos que ya se han extinguido y de los cuales quizás no tenemos evidencias físicas, no tenemos el esqueleto, pero podemos inferir su existencia a partir de los seres vivos actuales.
La historia que os querría explicar hoy va mucho más allá de los museos y de los fósiles que conocemos, pero os prometo que tiene de todo. Tiene un poco de intriga, una pizca de perseverancia y de intuición, tiene mucho de indagar en el pasado para inferir qué es lo que tenemos en la actualidad, y al mismo tiempo, abre puertas a futuras aplicaciones en la biomedicina o la biotecnología que todavía no hemos diseñado, ni siquiera imaginado. Os querría hablar de bacterias extintas y de resucitar genes y proteínas que ahora ya no existen, recreando estos paleogenes y paleoproteínas mediante análisis bioinformáticos e ingeniería genética, para darles vida de nuevo. Y para acabarlo de arreglar, esta investigación ha sido realizada mayoritariamente por compañeros científicos de España a los que conozco y admiro.
Primero me dejaréis explicaros el contexto, un contexto que tiene Premio Nobel detrás. Las bacterias tienen también sus parásitos, la mayoría son virus (que se llaman fagos o bacteriófagos, es decir "comedores de bacterias"). Las bacterias se tienen que defender de estos ataques que las fulminarían y, entre otros mecanismos, han incorporado un sistema para almacenar fotos del DNA de sus atacantes. Igual que la policía tiene un registro de fotos de los delincuentes con el fin de estar atentos y detectar su presencia, las bacterias incorporan fragmentos del material genético de sus virus y otros parásitos, con el fin de reconocerlos si los vuelven a atacar. Cuando un bacteriófago los penetra de nuevo, comparan el genoma del atacante con los fragmentos de genoma foto que tenían almacenados y si coinciden, los destruyen. ¿Y cómo lo hacen? Mediante un bisturí molecular con GPS, el GPS detecta que las fotos del DNA atacante y el almacenado son idénticas, y activan un sistema de corte del DNA atacante. Este sistema se llama CRISPR (el nombre que se refiere al sistema de almacenamiento y reconocimiento del DNA) y se suele acompañar del nombre del "bisturí" molecular, la enzima Cas9 (o de otros similares), por lo tanto, sistema CRISPR/Cas. Este sistema de reconocimiento y corte del DNA es actualmente usado como base del sistema de edición genética en todos los laboratorios del mundo, una herramienta imprescindible como lo son los cuchillos, tijeras, cuchillas y bisturíes de todo tipo en cualquier casa, y el número de patentes de variantes del sistema CRISPR y sus aplicaciones es inmenso. Pensad, por ejemplo, cuando modificáis cualquier texto, siempre utilizáis el buscador y el puntero del ratón para saber dónde vais y cómo recortáis, es decir, editáis buscando, cortando, revisando y pegando. Pues bien, el sistema CRISPR/Cas de edición genética fue descrito por Francis Mojica (un magnífico microbiólogo de Alacant, cuya historia os expliqué en otro artículo), aunque su uso como herramienta de edición genética fue premiado en el Premio Nobel de Química 2020 a Jennifer Doudna y Emmanuelle Charpentier (uno de los pocos Premios Nobel exclusivo de mujeres científicas).
Muchas de las respuestas del futuro las tenemos que buscar en organismos que ya se han extinguido y de los cuales quizás no tenemos evidencias físicas, no tenemos el esqueleto, pero podemos inferir su existencia a partir de los seres vivos actuales
Ahora bien, este sistema CRISPR/Cas de edición genética que sirve de defensa contra atacantes de las bacterias es muy y muy ancestral, ya que lo encontramos en especies bacterianas muy alejadas evolutivamente, y la diversidad que encontramos en cada especie refleja la selección natural. El sistema CRISPR/Cas que se utiliza más en los laboratorios deriva de bacterias relativamente comunes en nuestro hábitat, como Streptococcus piogenes o Staphilococcus aureus. Con el fin de buscar nuevos tipos y variantes del sistema para nuevas aplicaciones o mejoras, una opción frecuente es buscar especies bacterianas extremófilas que viven en lugares inhóspitos o de difícil acceso, por ejemplo, fuentes termales de los fondos abisales, o desiertos, como el de Atacama. En este caso, la diversidad bacteriana es espacial, pero podríamos pensar en traspasar la barrera del tiempo, yendo a buscarla en el pasado. ¿Cómo podemos buscar proteínas fósiles de especies bacterianas extinguidas hace millones de años? Recordad que hace poco os expliqué que se ha podido aislar DNA de depósitos minerales y rocas de hace 2 millones de años como un gran hito... pero podemos intentar inferir cuál es la secuencia más probable de genes y proteínas ancestrales (paleoproteínas) mediante la comparación de las especies bacterianas actuales con un análisis de parsimonia y, así, intentar inferir cuál tendría que ser la paleosecuencia más probable.
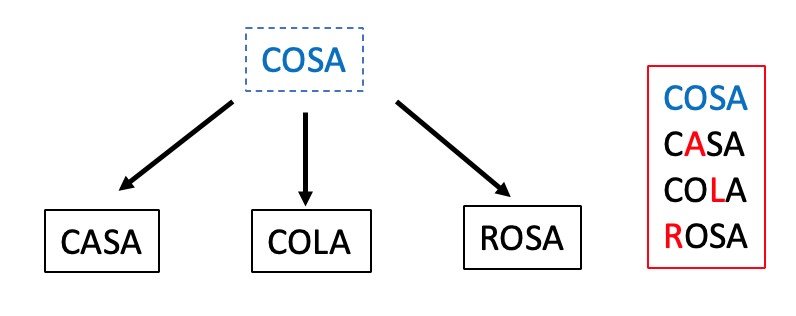
A continuación, os pongo un ejemplo sencillo de palabras muy parecidas, pero que todas difieren en una sola letra: CASA, COLA, ROSA. ¿Cuál sería la palabra común a todas ellas, a partir de la cual habrían evolucionado, solo cambiando una letra? Si las alineamos y comparamos, veréis que podemos proponer COSA como palabra original, ya que todas las otras se derivarían fácilmente solo cambiando la letra remarcada en rojo. Este ejemplo sencillo me permite ejemplarizar cómo se hacen los análisis bioinformáticos de alineamiento y de búsqueda de la palabra ancestral más parsimoniosa, la que necesitaría menos cambios para generar las otras tres palabras.
Así pues, los investigadores han hecho este análisis de inferencia para encontrar nuevas herramientas Cas (las enzimas que cortan el DNA), comparando especies bacterianas actuales de las cuales se calcula que divergieron de sus antepasados comunes hace 37, 137, 200, 1.000 y 2.600 millones de años. ¡Eso es increíble! Pensad que todos los dinosaurios se extinguieron hace unos 65 millones de años; que la explosión cámbrica, que dio lugar a la mayoría de clases animales que conocemos, tuvo lugar hace unos 580 millones de años; que la primera célula eucariota con núcleo, como las nuestras, surgió hace unos 1.850 millones de años, y que la vida en la Tierra tiene unos 3.500 millones de años de antigüedad... Sin embargo, los algoritmos bioinformáticos son capaces de inferir las secuencias de las proteínas paleo-Cas de hace 2.600 millones de años, las cuales se parecían a las actuales con más de un 56% de similitud, mientras que las paleo-Cas de hace 37 millones de años, se parecerían en un 93%. ¡No me digáis que no es espectacular!
Y ahora me podéis preguntar: ¿y cómo podemos saber si estas paleoproteínas predichas informáticamente existieron realmente? No lo podemos saber directamente, pero sí que podemos generarlas, resucitarlas mediante ingeniería genética. Si sabemos qué secuencia de aminoácidos tenían que tener, podemos generar sintéticamente estas proteínas y probar su actividad en el laboratorio, por ejemplo, sobre genes humanos. Como quizás ya intuís, estas proteínas sintéticas son eficientes al reconstituir el sistema CRISPR y pueden cortar el DNA. Cuanto más cerca en el tiempo, más eficientes y similares a las proteínas actuales son, lo cual parece lógico. Pero las más antiguas en el tiempo tienen actividades muy interesantes, seguramente adecuadas para un mundo en que no solo el DNA, sino también el RNA era la información genética predominante (para los que queréis saber un poco más, leed este fantástico artículo de divulgación de Lluís Montoliu, de uno de los científicos autores de este artículo).
Para acabar, o mejor dicho, para empezar, lo que os acabo de explicar sobre la resurrección de proteínas Cas ancestrales de hace millones de años implica un montón de perspectivas inexploradas con el fin de modificar estas nuevas herramientas de edición genética. Pensad en vuestro cajón de la cocina, en cuántos tipos de cuchillos diferentes tenéis... y ahora, pensad en cómo podremos aprender a utilizar nuevos tipos de cuchillos ancestrales que ni siquiera podíamos imaginar que existieron. ¡A mí me hacen chiribitas los ojos por todas las posibilidades y aplicaciones, en el futuro, de nuevos editores de texto genético en biotecnología y biomedicina, que ni siquiera habríamos pensado que fueran posibles!

