Fer trencaclosques és una activitat entretinguda. Hi ha trencaclosques de poques peces, amb els quals els nens petits juguen. Els ensenyem trucs que ajuden a resoldre’ls més fàcilment, com ara que les peces de les vores tenen un costat recte i que les peces de les cantonades són fàcilment distingibles. Quan hi ha poques peces, és fàcil anar reconstruint les figures, perquè sempre tenim algun detall que ens permet distingir la peça del trencaclosques que encaixa. Però, a mesura que busquem nous reptes, i el nombre de peces incrementa molt, costa molt més trobar els encaixos, i llavors, s’imposa separar primer les peces per colors predominants, i dins de cada muntet, per formes específiques, d’aquesta manera, simplifiquem la cerca de cada peça específica. La destresa en solucionar trencaclosques és, de fet, un conjunt de competències molt útils en el treball científic, perquè implica ordenació, classificació i reconeixement de patrons. Ens ordena mentalment. Convindreu amb mi, però, que alguns trencaclosques a partir de 1.000 peces són particularment difícils en algunes zones. Un cel uniformement blau que no el trenca cap núvol, un fragment de mar calm, o una escena d’un desert en què no hi ha dunes que projectin ombres reconeixibles ens suposen un repte superior i, moltes vegades, has d’anar provant peça a peça, fins que trobes la que quadra.
Doncs bé, el genoma humà que es va publicar el 2001 era un esborrany, però no era el genoma complet. Per poder-ho entendre, us diré que el genoma humà és com un trencaclosques gegant que vam seqüenciar a partir de milions de petites peces, que es van unir entre elles mitjançant patrons de seqüència compartida (evidentment, gràcies a programes bioinformàtics) per tal d’anar muntant tota la seqüència del genoma humà. Ja podeu imaginar que no va ser una tasca trivial. De fet, tenim regions cromosòmiques “fàcils”, en què hi ha seqüències molt específiques i úniques que ajuden a col·locar la peça sense dubtes, altres regions que tenim “ballant” una mica, i d’altres que senzillament han sigut impossibles de muntar perquè tècnicament no era possible. Imagineu-vos que teniu un trencaclosques de 10.000 peces, que ho teniu quasi tot muntat, i veieu perfectament la figura principal i molts dels detalls, però us han quedat uns buits a diferents zones, com unes 800 peces sense col·locar. De fet, així hem estat treballant fins ara amb el genoma, amb un 8% de la seva seqüència que no havíem pogut ni tan sols seqüenciar. Per què? Doncs perquè dins del nostre genoma tenim regions molt repetitives, en què la seqüència és molt similar (com si tinguéssiu 100 peces de cel blau, del mateix color i forma, i sense cap núvol). Evidentment que sí que sabíem on teníem els buits, però no sabíem com omplir-los, fins ara.
Aquesta setmana s’acaba de publicar un article a Science en què s’ha seqüenciat de manera completa, per primer cop, un genoma humà (fixeu-vos que diem “un” genoma, no pas “el” genoma), més sis altres articles relacionats que fan anàlisi de les regions que ara ja són conegudes. Si fins ara no s’havien pogut omplir els buits, com s’ha fet ara? Doncs utilitzant tècniques de seqüenciació massiva de lectures llargues, que permeten seqüenciar fragments més llargs del cromosoma. Seria com obtenir “peces més grans” del trencaclosques (entre 20 a 100 vegades més grans). Imagineu-vos que en lloc de tenir trossejat el genoma humà en les 10.000 peces que us he dit, ara la imatge del trencaclosques està continguda en només 1.000 peces. Com que les peces són molt més grans, ara és molt més fàcil fer el trencaclosques, perquè moltes de les peces difícils ara van juntes i trobem ja algun detall diferencial que ens permet inferir com van unides amb les altres. Els investigadors també han après que els calia minimitzar errors, i per això, han seqüenciat cèl·lules d’un embrió humà no viable originat per error, una mola hidatiforme, en què els 23 cromosomes s’han duplicat i són idèntics (això ha simplificat molt els possibles errors de seqüenciació i assignació). Aquest genoma complet, de punta a punta dels 23 cromosomes, de telòmer a telòmer (projecte T2T), té un cromosoma de cada parell de l’1 al 22, més el cromosoma X sencer. Aquest embrió venia d’una cèl·lula gamètica sense cromosoma Y i, per tant, aquest cromosoma encara no el tenim totalment seqüenciat.
Aquest genoma complet només és el primer de molts, ja que no conté tota la diversitat humana. Com que els humans estem formats per moltes poblacions d’origen diferent, es considera que com a mínim s’hauran de seqüenciar totalment fins a 350 genomes humans de persones d’origen diferent
On eren els forats que ens quedaven per seqüenciar? A les regions amb seqüències repetides, és a dir, als centròmers de cada cromosoma i als braços curts dels cromosomes 13, 14, 15 20 i 21. Fixeu-vos en la imatge que us adjunto, on es ressalten en vermell tots els “forats” del genoma humà que ara ja tenim plens.
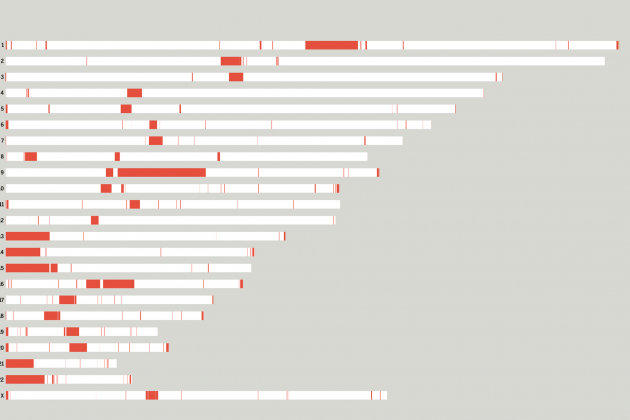
Cada barra blanca representa un cromosoma a escala, en vermell les seqüències que encara ens faltaven per cobrir / Imatge de V. Altounian amb les dades del Consorci T2T, extreta de doi: 10.1126/science.abp8653)
I ara em podeu preguntar: i és tan important el que hi ha codificat en aquest DNA? Quines conclusions en treuen? En primer lloc, per aconseguir aquesta fita, s’ha necessitat un equip multidisciplinari de molta gent i centres d’investigació dels Estats Units diferents. Van formar un consorci, anomenat T2T (de telòmer a telòmer), que té una pàgina web amb diversos vídeos. Jo us recomano especialment el primer, on una de les líders del projecte, Karen H. Miga, us explica en dos minuts quines són les fites més rellevants. També us recomano l’últim vídeo, d’animació, on de forma molt il·lustrativa i amena (professors, estudiants i gent encuriosida, paga la pena!) us explica que aquest genoma complet només és el primer de molts, ja que no conté tota la diversitat humana. Com que els humans estem formats per moltes poblacions d’origen diferent, es considera que com a mínim s’hauran de seqüenciar totalment fins a 350 genomes humans de persones d’origen diferent, és el projecte PanGenoma.
Per a qui us faci mandra anar a la pàgina web del T2T, us faré a continuació un molt breu resum perquè pugueu copsar la rellevància d’omplir tots aquests forats (a més de la satisfacció d’acabar el trencaclosques, és clar!):
1) Hem descobert que el nostre genoma té 3.055 milions de bases, 19.969 gens que codifiquen per proteïnes. En aquestes noves regions, n’hem descobert més de 1.956 de nous, que desconeixíem, dels quals 99 segurament són funcionals.
2) Aquestes regions són molt diverses genèticament, i estan relacionades amb malalties humanes. Ara podrem estudiar millor quines són les mutacions causatives.
3) Per fi, podem tenir la seqüència dels centròmers dels cromosomes humans, que conté una seqüència de DNA molt repetida (anomenada DNA satèl·lit alfa). Penseu que quan la cèl·lula replica, en aquest DNA s’hi uneixen els microtúbuls que estiren els cromosomes a les cèl·lules filles. Cada cèl·lula del nostre cos té 46 cromosomes (23 de cada progenitor), perquè aquestes seqüències són reconegudes correctament i fan de punt d’aterratge específic dels microtúbuls del fus acromàtic. Ara podem saber que aquestes seqüències dels centròmers, que pensàvem que eren totes iguals, són subtilment diferents entre algunes poblacions humanes.
4) En els cromosomes hi ha moltes regions riques en elements transponibles, seqüències que poden saltar i canviar de posició i que tot i tenir un origen “parasític”, van envair el genoma dels nostres ancestres més llunyans, ara són part integral del nostre genoma i són imprescindibles per a regular altres gens. Tanmateix, al ser repetides (prop de la meitat del nostre genoma són elements transponibles) ens duien a errors de localització, i ara podem estudiar millor quin és el seu efecte regulador del genoma.
5) Podem fer una anàlisi exhaustiva de la regulació epigenètica del nostre genoma, ara sí, sense que falti cap seqüència rellevant.
6) Podem estudiar molt millor la variabilitat humana, sobretot en regions que estan duplicades (on solen localitzar-se molts dels gens involucrats en la neurogènesi i implicats en diferents malalties i condicions psiquiàtriques).
7) Podem ara estudiar comparativament com el nostre genoma ha anat canviat durant l’evolució de la nostra espècie respecte a la d’altres simis i altres mamífers.
Ara només caldrà que tota la informació que teníem del genoma humà la puguem traspassar al nou genoma. Algunes coordenades han canviat, ja que teníem un mapa incomplet i ara passem a un de millor. Això és similar a quan ens comprem un ordinador amb un sistema operatiu millor, el traspàs d’informació no sempre és òptim i no tots els programes funcionen, almenys inicialment. Els que treballem en genètica humana ens hi haurem d’adaptar, però ningú vol treballar amb un ordinador antic quan en tens un de nou amb millors programes. Un genoma humà complet. Un gran avenç, sens dubte!

