Hacer puzles es una actividad entretenida. Hay puzles de pocas piezas, con los que juegan los niños pequeños. Les enseñamos trucos que ayudan a resolverlos más fácilmente, como que las piezas de los bordes tienen un lado recto y que las piezas de las esquinas son fácilmente distinguibles. Cuando hay pocas piezas, es fácil ir reconstruyendo las figuras, porque siempre tenemos algún detalle que nos permite distinguir la pieza del puzle que encaja. Sin embargo, a medida que buscamos nuevos retos, y el número de piezas incrementa mucho, cuesta mucho más encontrar los encajes, y entonces, se impone separar primero las piezas por colores predominantes, y dentro de cada montoncito, por formas específicas, de esta manera, simplificamos la búsqueda de cada pieza específica. La destreza al solucionar puzles es, de hecho, un conjunto de competencias muy útiles en el trabajo científico, porque implica ordenación, clasificación y reconocimiento de patrones. Nos ordena mentalmente. Estaréis de acuerdo conmigo, sin embargo, en que algunos rompecabezas a partir de 1.000 piezas son particularmente difíciles en algunas zonas. Un cielo uniformemente azul que no lo rompe ninguna nube, un fragmento de mar en calma, o una escena de un desierto en el que no hay dunas que proyecten sombras reconocibles nos suponen un reto superior y, muchas veces, tienes que ir probando pieza a pieza, hasta que encuentras la que cuadra.
Pues bien, el genoma humano que se publicó en 2001 era un borrador, pero no era el genoma completo. Para poder entenderlo, os diré que el genoma humano es como un puzle gigante que secuenciamos a partir de millones de pequeñas piezas, que se unieron entre ellas mediante patrones de secuencia compartida (evidentemente, gracias a programas bioinformáticos) con el fin de ir montando toda la secuencia del genoma humano. Ya os podéis imaginar que no fue una tarea trivial. De hecho, tenemos regiones cromosómicas "fáciles", en las que hay secuencias muy específicas y únicas que ayudan a colocar la pieza sin dudas, otras regiones que tenemos "bailando" un poco, y otras que sencillamente han sido imposibles de montar porque técnicamente no era posible. Imaginaos que tenéis un rompecabezas de 10.000 piezas, que lo tenéis casi todo montado, y veis perfectamente la figura principal y muchos de los detalles, pero os han quedado unos huecos en diferentes zonas, como unas 800 piezas sin colocar. De hecho, así hemos estado trabajando hasta ahora con el genoma, con un 8% de su secuencia que no habíamos podido ni siquiera secuenciar. ¿Por qué? Pues porque dentro de nuestro genoma tenemos regiones muy repetitivas, en las que la secuencia es muy similar (como si tuvierais 100 piezas de cielo azul, del mismo color y forma, y sin ninguna nube). Evidentemente que sí sabíamos dónde teníamos los huecos, pero no sabíamos cómo llenarlos, hasta ahora.
Esta semana se acaba de publicar un artículo en Science en el que se ha secuenciado de manera completa, por primera vez, un genoma humano (fijaos en que decimos "un" genoma, no "el" genoma), más seis otros artículos relacionados que hacen análisis de las regiones que ahora ya son conocidas. Si hasta ahora no se habían podido llenar los huecos, ¿cómo se ha hecho ahora? Pues utilizando técnicas de secuenciación masiva de lecturas largas, que permiten secuenciar fragmentos más largos del cromosoma. Sería como obtener "piezas grandes" del rompecabezas (entre 20 a 100 veces más grandes). Imaginaos que en lugar de tener troceado el genoma humano en las 10.000 piezas que os he dicho, ahora la imagen del puzle está contenida en sólo 1.000 piezas. Como las piezas son mucho más grandes, ahora es mucho más fácil hacer el rompecabezas, porque muchas de las piezas difíciles ahora van juntas y encontramos ya algún detalle diferencial que nos permite inferir cómo van unidas con las demás. Los investigadores también han aprendido que tenían que minimizar errores, y por eso, han secuenciado células de un embrión humano no viable originado por error, una mole hidatiforme, en el que los 23 cromosomas se han duplicado y son idénticos (eso ha simplificado mucho los posibles errores de secuenciación y asignación). Este genoma completo, de punta a punta de los 23 cromosomas, de telómero a telómero (proyecto T2T), tiene un cromosoma de cada par del 1 al 22, más el cromosoma X entero. Este embrión venía de una célula gamética sin cromosoma Y y, por lo tanto, este cromosoma todavía no lo tenemos totalmente secuenciado.
Este genoma completo sólo es el primero de muchos, ya que no contiene toda la diversidad humana. Como los humanos estamos formados por muchas poblaciones de origen diferente, se considera que como mínimo se tendrán que secuenciar totalmente hasta 350 genomas humanos de personas de origen diferente
¿Dónde estaban los agujeros que nos quedaban por secuenciar? En las regiones con secuencias repetidas, es decir, en los centrómeros de cada cromosoma y en los brazos cortos de los cromosomas 13, 14, 15 20 y 21. Fijaos en la imagen que os adjunto, donde se resaltan en rojo todos los "agujeros" del genoma humano que ahora ya hemos llenado.
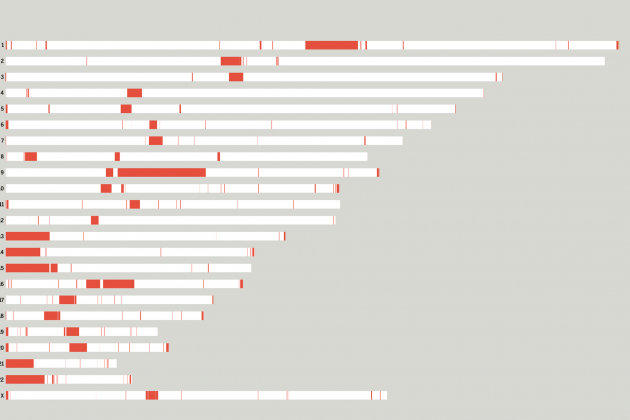
Cada barra blanca representa un cromosoma a escala, en rojo las secuencias que todavía nos faltaban por cubrir / Imagen de V. Altounian con los datos del Consorcio T2T, extraída de doi: 10.1126/science.abp8653)
Y ahora me podéis preguntar: ¿y es tan importante lo que hay codificado en este DNA? ¿Qué conclusiones extraen? En primer lugar, para conseguir este hito, se ha necesitado un equipo multidisciplinar de mucha gente y centros de investigación de los Estados Unidos diferentes. Formaron un consorcio, denominado T2T (de telómero a telómero), que tiene una página web con varios vídeos. Yo os recomiendo especialmente el primero, donde una de las líderes del proyecto, Karen H. Miga, os explica en dos minutos cuáles son los hitos más relevantes. También os recomiendo el último vídeo, de animación, en el que de forma muy ilustrativa y amena (profesores, estudiantes y gente interesada, ¡vale la pena!) os explica que este genoma completo sólo es el primero de muchos, ya que no contiene toda la diversidad humana. Como los humanos estamos formados por muchas poblaciones de origen diferente, se considera que como mínimo se tendrán que secuenciar totalmente hasta 350 genomas humanos de personas de origen diferente, es el proyecto PanGenoma.
Para quien le dé pereza ir a la página web del T2T, os haré a continuación un muy breve resumen para que podáis asimilar la relevancia de llenar todos estos agujeros (¡además de la satisfacción de acabar el rompecabezas, claro!):
1) Hemos descubierto que nuestro genoma tiene 3.055 millones de bases, 19.969 genes que codifican para proteínas. En estas nuevas regiones, hemos descubierto más de 1.956 nuevos, que desconocíamos, de los cuales 99 seguramente son funcionales.
2) Estas regiones son muy diversas genéticamente, y están relacionadas con enfermedades humanas. Ahora podremos estudiar mejor cuáles son las mutaciones causativas.
3) Por fin, podemos tener la secuencia de los centrómeros de los cromosomas humanos, que contiene una secuencia de DNA muy repetida (denominada DNA satélite alfa). Pensad que cuando la célula replica, a este DNA se le unen los microtúbulos que estiran los cromosomas a las células hijas. Cada célula de nuestro cuerpo tiene 46 cromosomas (23 de cada progenitor), porque estas secuencias son reconocidas correctamente y hacen de punto de aterrizaje específico de los microtúbulos del huso acromático. Ahora podemos saber que estas secuencias de los centrómeros, que pensábamos que eran todos iguales, son sutilmente diferentes entre algunas poblaciones humanas.
4) En los cromosomas hay muchas regiones ricas en elementos transponibles, secuencias que pueden saltar y cambiar de posición y que a pesar de tener un origen "parasítico", invadieron el genoma de nuestros ancestros más lejanos, ahora son parte integral de nuestro genoma y son imprescindibles para regular otros genes. Sin embargo, al ser repetidas (cerca de la mitad de nuestro genoma son elementos transponibles) nos llevaban a errores de localización, y ahora podemos estudiar mejor cuál es su efecto regulador del genoma.
5) Podemos hacer un análisis exhaustivo de la regulación epigenética de nuestro genoma, ahora sí, sin que falte ninguna secuencia relevante.
6) Podemos estudiar mucho mejor la variabilidad humana, sobre todo en regiones que están duplicadas (donde suelen localizarse muchos de los genes involucrados en la neurogénesis e implicados en diferentes enfermedades y condiciones psiquiátricas).
7) Podemos ahora estudiar comparativamente cómo nuestro genoma ha ido cambiado durante la evolución de nuestra especie con respecto a la de otros simios y otros mamíferos.
Ahora sólo faltará que toda la información que teníamos del genoma humano la podamos traspasar al nuevo genoma. Algunas coordenadas han cambiado, ya que teníamos un mapa incompleto y ahora pasamos a uno mejor. Eso es similar a cuando nos compramos un ordenador con un sistema operativo mejor, el traspaso de información no siempre es óptimo y no todos los programas funcionan, al menos inicialmente. Los que trabajamos en genética humana nos tendremos que adaptar, pero nadie quiere trabajar con un ordenador antiguo cuando tienes uno nuevo con mejores programas. Un genoma humano completo. ¡Un gran avance, sin duda!

