Tots coneixem la figura dels gnoms. Es troben a molts contes infantils, com a nans irascibles que viuen al bosc, molt vinculats a la natura i a les mines, que sovint guarden tresors d'or i pedres precioses. Fins i tot els visualitzem amb una espècie de barretina punxeguda de color vermell o verd, sota un bolet vermell amb taques blanques (el reig bord o Amanita muscaria, que, per cert, és un bolet molt tòxic i no se us ha d'ocórrer mai de menjar-lo). Però hem d'admetre que els gnoms són personatges de conte que, en general, ens cauen simpàtics.
Els anglosaxons usen moltes vegades els jocs de paraules fonètics i els acrònims per donar nom a conceptes nous. En ciència és molt útil, perquè ens ajuda a recordar el nom d'arxius, de convocatòries, de programes d'ordinador, de mutacions o de gens, de forma més fàcil i amable. Així, doncs, no us estranyarà que un banc de dades del genoma complet de moltes persones es digui gnomAD (de genome aggregation database). Pels qui fem servir aquest nom, ens ve al cap immediatament la imatge d'un gnom. Aquest banc de dades del genoma és la continuació natural d'un altre banc de dades de seqüències d'ADN humà, un banc de dades de tots els gens (l'exoma), anomenat ExAC, que ens recorda al mot "exacte". Aquí cal apuntar que el nostre genoma és molt més gran del que pensaríem a priori, ja que menys d'un 2% del nostre genoma codifica per a gens (de fet, encara estem intentant esbrinar quina és la funció de bona part del 98% restant). Per tant, l'anàlisi de l'exoma (més fàcil i concret) només estudia el 2 % de tot el nostre ADN.
Així, doncs, entre el gnom i l'exacte, tenim una gran quantitat d'informació genètica dels éssers humans i de la variabilitat que presentem entre nosaltres. Pels que ens dediquem a la genètica humana, ja sempre més recordarem que, enmig de la gran rellevància de la pandèmia de la Covid-19, s'ha fet un avenç indiscutible i molt necessari en el coneixement de què som, els humans, i per què som diversos entre nosaltres, ja que s'han actualitzat tots dos bancs de dades gràcies a l'esforç ingent de més de 100 investigadors que han seqüenciat 125.748 exomes (la part de la nostra informació genètica que codifica per gens) i 15.708 genomes, que s'afegeixen a la informació que ja teníem. Estem parlant de més de 3 petabytes d'informació; peta- és un prefix que vol dir 10 elevat a 15, perquè us en feu una idea, un gigabyte són 1.000 milions de bytes, és a dir, 10 elevat a 9. Així, doncs, estem parlant de tres milions de vegades més que la informació d'un gigabyte. Imagineu de quants llapis de memòria estem parlant!
Aquesta informació és crucial per poder comprendre com i per què tenim malalties genètiques, i aquesta informació ens servirà en un futur per dissenyar millors medicaments per a curar o pal·liar els seus efectes
I ara que ja us he marejat una mica amb les xifres, em podeu preguntar per què als genetistes ens sembla tan important que s'hagin publicat tot un seguit d'articles a les millors revistes del camp explicant els avenços que s'han fet. Doncs perquè aquesta informació és crucial per poder comprendre com i per què tenim malalties genètiques, i aquesta informació ens servirà en un futur per dissenyar millors medicaments per a curar o pal·liar els seus efectes.
Aquesta nova informació ens diu que, de moment, s'han identificat més de 241 milions de variants genètiques d'una sola posició, és a dir, de "lletres" de l'ADN que poden ser diferents en els humans, un gran avenç si tenim en compte que abans només en coneixíem poc més de 7 milions, però aquestes variants afecten normalment els gens que codifiquen proteïnes. Però, a més, hem descobert que podem tenir més de 335.000 variants estructurals, que vindrien a ser paràgrafs sencers canviats de lloc, duplicats o directament esborrats) del llibre d'instruccions que és el nostre genoma. I tot aquest catàleg de variació humana està a disposició pública, ja que el gnomAD i l'ExAC són bancs de dades oberts a tothom.
Quan fem diagnòstic genètic, sigui perquè estem treballant amb pacients de malalties rares i cal esbrinar quina és la informació genètica que està mutada, amb pacients que tenen un trastorn de l'espectre autista i volem saber quina és la raó genètica, amb pacients que tenen càncer i volem conèixer quin és el gen de predisposició, o bé quan voldrem predir si una persona serà més o menys susceptible a patir conseqüències molt greus si s'infecta pel SARS-CoV-2, hem d'analitzar el seu ADN i tractar de trobar aquestes variants genètiques causatives o de risc. El que fem els genetistes és comparar l'ADN del pacient amb l'ADN del banc de dades i les variants ja descrites, per tal de fer inferències sobre quina és la instrucció genètica diferent que pot ser la causativa de la malaltia. És com si juguéssim al joc de les 7 diferències. Si mai hi heu jugat, sabreu que teniu dues imatges quasi idèntiques i cal trobar en quins punts les dues imatges són lleugerament diferents. Nosaltres fem això amb tots els gens humans quan fem un diagnòstic genètic, però cal que tinguem un banc de dades molt complet per poder esbrinar quina de totes les variants que hem trobat és la causativa. Hem de filar molt prim, i com més dades genètiques de moltes persones de referència tinguem, amb major precisió podrem establir relacions de causalitat.
Un dels resultats més interessants és veure que hi ha gens, instruccions genètiques, que són redundants. Resulta que hi ha persones, considerades "normals", és a dir, persones sense cap afectació clínica evident, però que, tot i això, són portadors de mutacions importants, de pèrdua de funció, en totes dues còpies (l'heretada per part de pare i l'heretada per part de mare) del mateix gen. De moment, han identificat 1.815 gens que podrien ser "no necessaris". I a l'inrevés, han detectat gens vitals, en els quals no hi ha variants genètiques inactivadores, molt probablement perquè si les tinguéssim, patiríem una malaltia greu o, fins i tot, perquè les mutacions a les dues còpies heretades comporten letalitat, és a dir, la mort de la persona. També es poden trobar variants genètiques de disminució de la funció d'un gen sense implicar malaltia, i ens pot indicar, doncs, a quin lloc de la proteïna cal dirigir un nou medicament perquè pugui ser efectiu sense excessius efectes secundaris, per exemple, amb el gen LRRK2, associat a la malaltia de Parkinson familiar.
Per últim, els investigadors comenten que més de la meitat dels genomes que han analitzat són de persones europees (per exemple, Finlàndia, Suècia, Estònia i el Regne Unit han contribuït amb els seus bancs públics d'ADN), encara que també hi ha ADN de persones d'origen asiàtic i jueus asquenazites (mireu l'esquema de colors adjunt), però, clarament, manca la informació genètica d'altres poblacions, com ara les indígenes de certes regions geogràfiques aïllades, i també, de moltes poblacions africanes. No oblidem que és de les poblacions africanes que esperem una major diversitat genètica humana, atès que els humans hem sorgit a l'Àfrica.
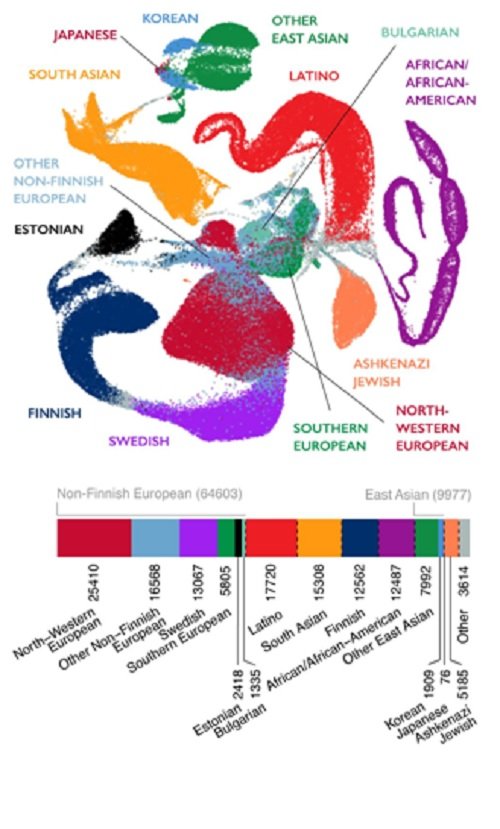
Cada punt representa l'ADN seqüenciat d'una persona, amb un codi de colors segons el seu origen genètic-geogràfic (Figura obtinguda del projecte gnomAD, segons publicacions a Nature, 28 de maig de 2020)
Tot això ens indica que s'ha fet un gran avenç en l'estudi de la variabilitat genètica humana, però que encara ens queda molta més variació humana per estudiar. Queden molts gnoms per estudiar!

