Todos conocemos la figura de los gnomos. Salen en muchos cuentos infantiles, como enanos irascibles que viven en el bosque, muy vinculados a la naturaleza y a las minas, que a menudo guardan tesoros de oro y piedras preciosas. Incluso los visualizamos con una especie de gorro puntiagudo de color rojo o verde, bajo una seta roja con manchas blancas (la falsa oronja o Amanita muscaria, que, por cierto, es una seta muy tóxica y no se os ocurra nunca comerla). Pero tenemos que admitir que los gnomos son personajes de cuento que, en general, nos caen simpáticos.
Los anglosajones usan muchas veces los juegos de palabras fonéticos y los acrónimos para dar nombre a conceptos nuevos. En ciencia es muy útil, porque nos ayuda a recordar el nombre de archivos, de convocatorias, de programas de ordenador, de mutaciones o de genes, de forma más fácil y amable. Así, pues, no os extrañará que un banco de datos del genoma completo de muchas personas se llame gnomAD (de genome aggregation database). A los que utilizamos este nombre, nos viene a la cabeza inmediatamente la imagen de un gnomo. Este banco de datos del genoma es la continuación natural de otro banco de datos de secuencias de ADN humano, un banco de datos de todos los genes (el exoma), llamado ExAC, que nos recuerda a la palabra "exacto". Aquí hay que precisar que nuestro genoma es mucho más grande de lo que pensaríamos a priori, ya que menos de un 2% de nuestro genoma codifica para genes (de hecho, todavía estamos intentando averiguar cuál es la función de gran parte del 98% restante). Por lo tanto, el análisis del exoma (más fácil y concreto) sólo estudia el 2% de todo nuestro ADN.
Así, pues, entre el gnomo y el exacto, tenemos una gran cantidad de información genética de los seres humanos y de la variabilidad que presentamos entre nosotros. Para los que nos dedicamos a la genética humana, recordaremos para siempre que, en medio de la gran relevancia de la pandemia de la Covid-19, se ha hecho un avance indiscutible y muy necesario en el conocimiento de qué somos, los humanos, y por qué somos diversos entre nosotros, ya que se han actualizado los dos bancos de datos gracias al esfuerzo ingente de más de 100 investigadores que han secuenciado 125.748 exomas (la parte de nuestra información genética que codifica para genes) y 15.708 genomas, que se añaden a la información que ya teníamos. Estamos hablando de más de 3 petabytes de información; peta- es un prefijo que quiere decir 10 elevado a 15, para que os hagáis una idea, un gigabyte son 1.000 millones de bytes, es decir, 10 elevado a 9. Así, pues, estamos hablando de tres millones de veces más que la información de un gigabyte. ¡Imaginad de cuántos lápices de memoria estamos hablando!
Esta información es crucial para poder comprender cómo y por qué tenemos enfermedades genéticas, y esta información nos servirá en un futuro para diseñar mejores medicamentos para curar o paliar sus efectos
Y ahora que ya os he mareado un poco con las cifras, me podéis preguntar por qué a los genetistas nos parece tan importante que se hayan publicado toda una serie de artículos en las mejores revistas del campo explicando los avances que se han hecho. Pues porque esta información es crucial para poder comprender cómo y por qué tenemos enfermedades genéticas, y esta información nos servirá en un futuro para diseñar mejores medicamentos para curar o paliar sus efectos.
Esta nueva información nos dice que, de momento, se han identificado más de 241 millones de variantes genéticas de una sola posición, es decir, de "letras" del ADN que pueden ser diferentes en los humanos, un gran avance si tenemos en cuenta que antes sólo conocíamos poco más de 7 millones, pero estas variantes afectan normalmente a los genes que codifican proteínas. Sin embargo, además, hemos descubierto que podemos tener más de 335.000 variantes estructurales, que vendrían a ser párrafos enteros cambiados de lugar, duplicados o directamente borrados) del libro de instrucciones que es nuestro genoma. Y todo este catálogo de variación humana está a disposición pública, ya que el gnomAD y el ExAC son bancos de datos abiertos a todo el mundo.
Cuando hacemos diagnóstico genético, sea porque estamos trabajando con pacientes de enfermedades raras y hay que averiguar cuál es la información genética que está mutada, con pacientes que tienen un trastorno del espectro autista y queremos saber cuál es la razón genética, con pacientes que tienen cáncer y queremos conocer cuál es el gen de predisposición, o bien cuando querremos predecir si una persona será más o menos susceptible a sufrir consecuencias muy graves si se infecta por el SARS-CoV-2, tenemos que analizar su ADN y tratar de encontrar estas variantes genéticas causativas o de riesgo. Lo que hacemos los genetistas es comparar el ADN del paciente con el ADN del banco de datos y las variantes ya descritas, con el fin de hacer inferencias sobre cuál es la instrucción genética diferente que puede ser la causativa de la enfermedad. Es como si jugáramos al juego de las 7 diferencias. Si habéis jugado alguna vez, sabréis que tenéis dos imágenes casi idénticas y hay que encontrar en qué puntos las dos imágenes son ligeramente diferentes. Nosotros hacemos eso con todos los genes humanos cuando hacemos un diagnóstico genético, pero es necesario que tengamos un banco de datos muy completo para poder averiguar cuál de todas las variantes que hemos encontrado es la causativa. Tenemos que precisar mucho, y cuantos más datos genéticos de muchas personas de referencia tengamos, con mayor precisión podremos establecer relaciones de causalidad.
Uno de los resultados más interesantes es ver que hay genes, instrucciones genéticas, que son redundantes. Resulta que hay personas, consideradas "normales", es decir, personas sin ninguna afectación clínica evidente, pero que, sin embargo, son portadoras de mutaciones severas, de pérdida de función, en las dos copias (la heredada por parte de padre y la heredada por parte de madre) del mismo gen. De momento, han identificado 1.815 genes que podrían ser "no necesarios". Y al revés, han detectado genes vitales, en los cuales no hay variantes genéticas inactivadoras, muy probablemente porque si las tuviéramos, sufriríamos una enfermedad grave o, incluso, porque las mutaciones en las dos copias heredadas comportan letalidad, es decir, la muerte de la persona. También se pueden encontrar variantes genéticas de disminución de la función de un gen sin implicar enfermedad, y nos puede indicar, pues, a qué lugar de la proteína hay que dirigir un nuevo medicamento para que pueda ser efectivo sin excesivos efectos secundarios, por ejemplo, con el gen LRRK2, asociado a la enfermedad de Parkinson familiar.
Por último, los investigadores comentan que más de la mitad de los genomas que han analizado son de personas europeas (por ejemplo, Finlandia, Suecia, Estonia y el Reino Unido han contribuido con sus bancos públicos de ADN), aunque también hay ADN de personas de origen asiático y judíos asquenazíes (mirad el esquema de colores adjunto), sin embargo, claramente, falta la información genética de otras poblaciones, como las indígenas de ciertas regiones geográficas aisladas, y también, de muchas poblaciones africanas. No olvidemos que es de las poblaciones africanas que esperamos una mayor diversidad genética humana, dado que los humanos hemos surgido en África.
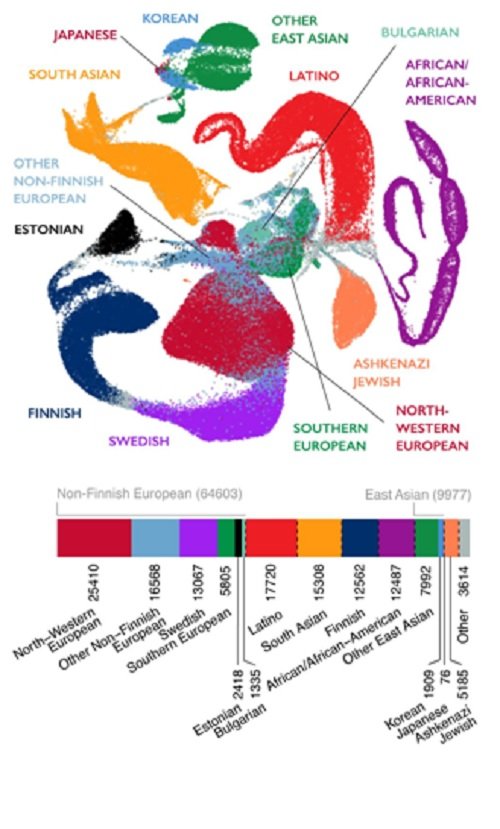
Cada punto representa el ADN secuenciado de una persona, con un código de colores según su origen genético-geográfico (Figura obtenida del proyecto gnomAD, según publicaciones en Nature, 28 de mayo de 2020)
Todo eso nos indica que se ha hecho un gran avance en el estudio de la variabilidad genética humana, pero que todavía nos queda mucha más variación humana por estudiar. ¡Quedan muchos gnomos por estudiar!

