Uno de los problemas sanitarios más graves es la falta de antibióticos efectivos contra muchas infecciones bacterianas. Se calcula que en torno a 10 millones de personas mueren en el mundo a causa de infecciones bacterianas no tratables. No es que no tengamos antibióticos, tenemos una buena colección de compuestos —inicialmente, naturales y, posteriormente, obtenidos sintéticamente— que cuando fueron purificados o diseñados eran muy activos contra una gran variedad de bacterias, pero su uso excesivo ha causado la selección de cepas bacterianas muy resistentes. La mayoría de antibióticos van dirigidos contra la pared bacteriana (una especie de muralla protectora exterior de las bacterias Gram+) o contra el metabolismo de la producción de proteínas de las bacterias. Muchas empresas farmacéuticas han desistido de buscar nuevos compuestos antibióticos porque la tarea es cada vez más pesada, para un éxito muy limitado en el tiempo. Sin embargo, hay aproximaciones totalmente innovadoras y creativas fuera del esquema habitual de identificación de nuevos productos con actividad antibacteriana.
Un grupo de investigación de los Estados Unidos, liderado por un investigador gallego, César de la Fuente, propone una visión totalmente rompedora para la búsqueda de nuevos compuestos con actividad antibiótica. Su hipótesis es que, aparte del sistema inmunitario, hay muchas moléculas proteicas dentro de nuestro cuerpo con potencial antibiótico, ya que muchas de estas proteínas circulan en sangre, o ejercen su función fuera de la célula, en un medio que puede estar infectado con bacterias, y que han podido ser seleccionadas para ejercer un efecto protector sobre el organismo, actuando como una defensa antibiótica contra ciertos elementos bacterianos, por ejemplo, rompiendo o desestabilizando su membrana plasmática. Esta acción antibacteriana estaría ejercida por algún dominio peptídico (una parte pequeña de la proteína pequeña, de solo 8 a 50 aminoácidos), inmerso dentro de la proteína grande, y habría sido seleccionada durante millones de años para hacer bien esta función. Si hacemos un símil sencillo, vendría a ser como una persona que lleva un arma de fuego o un puñal como herramienta de defensa (aunque la tarea de la persona pueda ser desde conductor de diligencias, un campesino o un ingeniero). El trabajo principal de la proteína es uno determinado, diferente del de este pequeño péptido de defensa, que se encuentra críptico, como "escondido". La cuestión es cómo "identificar" estos posibles péptidos antibióticos que están escondidos.
Quizás en un futuro no muy lejano, tomaremos una píldora con un péptido desextinguido del tiranosaurio para tratar la migraña, o un jarabe con un componente activo desextinguido de neandertal para tratar la osteoporosis
Con esta idea tan innovadora, este grupo de investigación desarrolló un algoritmo informático de inteligencia artificial de aprendizaje profundo (machine learning) que han entrenado para detectar nuevos antibióticos usando exclusivamente las propiedades fisicoquímicas de los péptidos. De este modo, detectaron más de 2.600 péptidos "encriptados" dentro del proteoma humano (el conjunto de proteínas que nuestro DNA codifica) que tienen la capacidad de matar bacterias que causan infecciones, rompiendo la membrana bacteriana, e incluso, pueden modular las bacterias comensales de nuestro intestino y nuestra piel. No todos estos péptidos son mejores que los antibióticos que ya tenemos, hay que distinguir los mejores, pero ya nos da una idea del potencial de esta aproximación. Evidentemente, los investigadores no se han detenido en este punto, y se han atrevido a dar un paso más allá. Si dentro del proteoma de los humanos actuales encontramos todos estos potenciales "antibióticos", quizás si escrutamos y analizamos el paleoproteoma de humanos extintos, como los neandertales y los denisovanos, también encontraremos nuevos, ya que sus proteínas son en algunos casos diferentes de las nuestras y también hacían frente a infecciones bacterianas diferentes. Dicho y hecho, una nueva versión del algoritmo de inteligencia artificial se ha usado para analizar las proteínas codificadas por el genoma de estos homínidos próximos a nosotros, con el fin de encontrar nuevos péptidos con capacidad antibiótica, y resucitarlos. Mirad la figura que resume este trabajo, recién publicado.
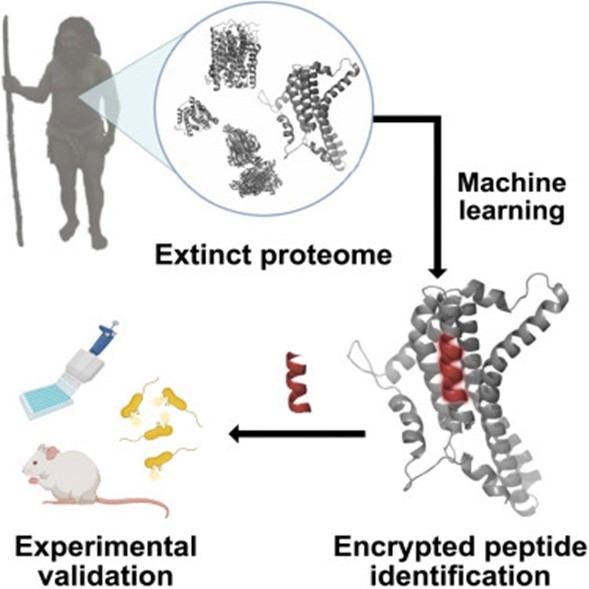
Tanto los péptidos antibióticos encriptados arcaicos (AEP) como los modernos (MEP) han sido seleccionados y escogidos, de modo que los que parecían mejores se han comprobado sobre infecciones bacterianas en la piel y el músculo del muslo en modelos de ratón, demostrando una eficacia similar a la de conocidos antibióticos actuales, con una citotoxicidad baja para el ratón y, de momento, con poca capacidad de generar bacterias resistentes.
Ahora bien, como las técnicas de secuenciación masiva han permitido secuenciar muchísimas especies, tanto de las que están actualmente vivas como extintas, existen un montón de datos genómicos por estudiar. Así que en un nuevo artículo (que todavía no está publicado en ninguna revista, sino que solo está depositado y es accesible), una mejora del algoritmo de aprendizaje profundo, ahora denominado APEX (del inglés Antibiotic Peptide Des-extinction, o desextinción de péptidos con actividad antibiótica), ha analizado este "extintoma" (el proteoma de todos los animales extinguidos, identificando aproximadamente 37.000 péptidos diferentes, los cuales han sido seleccionados, desextinguidos y comprobados, primero sobre cultivos celulares y, finalmente, se ha estudiado la efectividad de siete de ellos para controlar las infecciones en ratones. Estos péptidos-antibióticos tienen nombres derivados de los organismos extintos de los que se han obtenido y resucitado, como hydrodamina-1 (de la vaca marina extinta Hydradamalis gigas), la mylodonina-2 (del perezoso gigante Myodon darwinii) o la mammuthsina-2 (del mamut, Mammuthus primigenius).
Y evidentemente, esto es solo el principio, ya que también se pueden entrenar algoritmos para encontrar péptidos que tengan efectividad contra otras enfermedades que nos afectan, que no son infecciosas. ¿Quién sabe? Quizás en un futuro no muy lejano, tomaremos una píldora con un péptido desextinguido del tiranosaurio para tratar la migraña, o un jarabe con un componente activo desextinguido de neandertal para tratar la osteoporosis, o la tuberculosis. Inspirándose en el pasado para encontrar los medicamentos del futuro.

